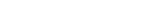“데이터는 이미 엄청나게 쌓여 있습니다. 어떤 분석 전략을 세우느냐에 따라 얼마든지 중요한 발견을 할 수 있고, 세포와 질병에 대한 이해도 높일 수 있어요.”
남덕우 생명과학부 교수팀은 ‘유전자 발현 빅데이터’ 분석을 통해 암을 억제하는 마이크로RNA와 이와 관련된 세포 신호조절 경로를 발굴했다. 마이크로RNA는 19~23개 정도의 짧은 염기로 이루어진 RNA 분자로서, 여러 유전자의 발현을 억제한다. 이를 통해 다양한 세포 활동과 암과 당뇨 등의 만성질환에 핵심적인 역할을 한다.
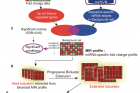
남 교수팀은 15년 이상 차곡차곡 쌓인 유전자 발현(gene expression) 공공 데이터베이스를 활용하는 새로운 분석 전략을 개발했다. 연구팀은 이 데이터베이스에서 각종 질병과 조직 특성, 세포 분화, 약물처리 등 다양한 세포 조건에 따른 5000여 개의 데이터 세트를 가공해 ‘유전자 발현 빅데이터’를 수집했다. 또 마이크로RNA의 염기서열에 기반한 타깃 유전자(target gene) 집단의 정보를 함께 분석했다. 그 결과, 459개의 ‘인간 마이크로RNA에 의한 조절 네트워크’를 예측하는 빅데이터 분석 시스템(BiMIR)을 구축할 수 있었다. (BiMIR 바로가기)
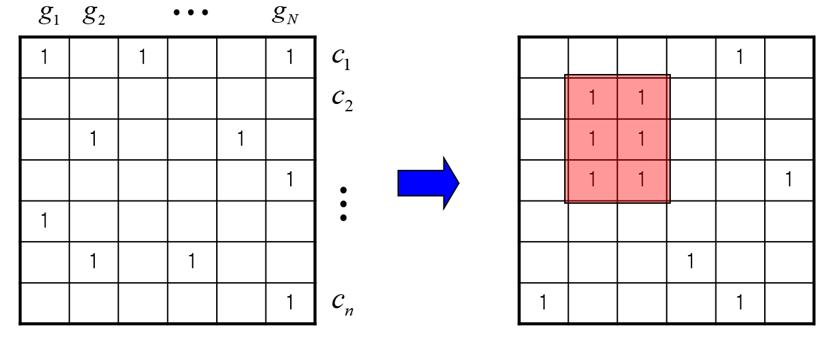
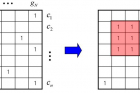
바이클러스터링 문제. 유전자 g와 세포 조건 c를 동시에 치환해 1로 가득한 블록을 찾는 문제다. 해당 블록에 포함되는 유전자들은 해당 세포 조건에서 동시에 발현되는 타깃 유전자들이다. 두 조건(bi)을 모두 만족하는 묶음(cluster)를 찾아낸다는 뜻에서 바이클러스터링이라는 이름이 붙었다.
특히 ‘바이클러스터링(Biclustering)’이라는 양방향 군집화 분석을 통해, 마이크로RNA가 조절하는 ‘유전자 집단’과 관련 ‘세포 조건’을 동시에 제시해주는 새로운 접근법을 개발했다.
남덕우 교수는 “유전자 발현 빅데이터에 바이클러스터링 방법을 적용하면, 줄기세포나 특정 질병 등 다양한 세포 조건에서 일어나는 마이크로RNA 조절 네트워크를 더 정확하게 발굴할 수 있다”며 “가령 유방암이 어떤 유전자들의 발현과 연결돼 있고, 이들 유전자를 억제하는 마이크로RNA가 무엇인지 예측하게 되는 것”이라고 설명했다.
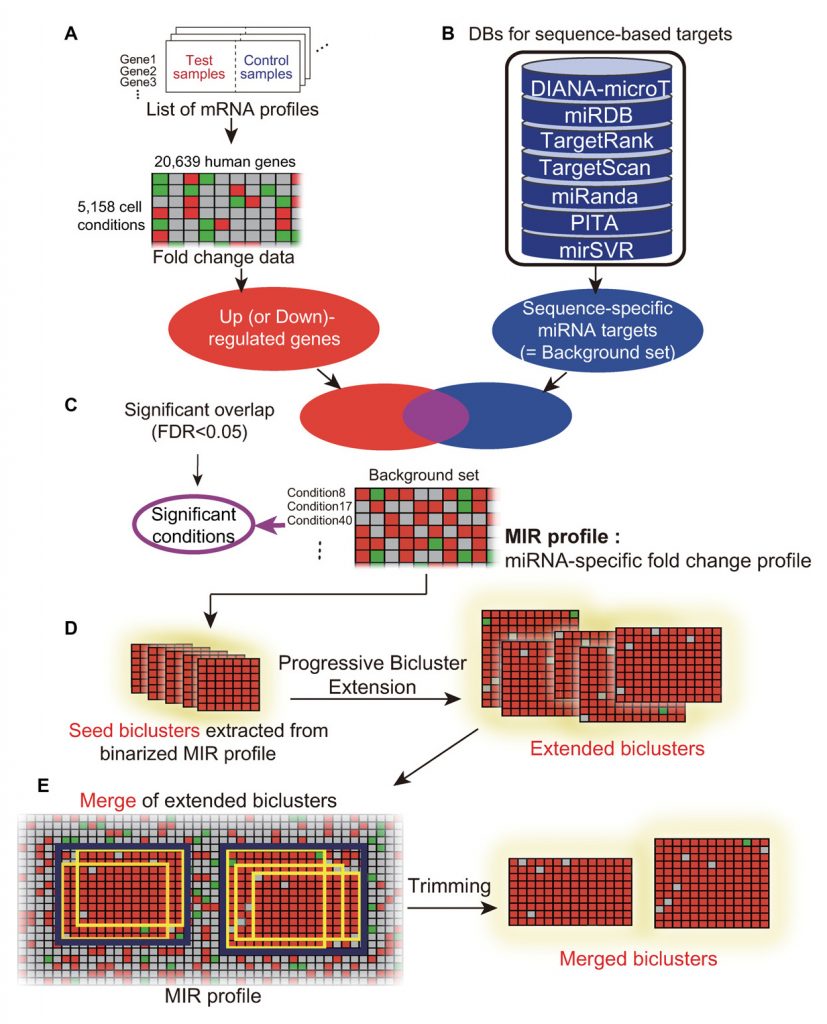
연구진은 실제로 유방암 발달에 중요한 신호전달 경로(PI3K/Akt signaling pathway)를 miR-29 등 적은 수의 마이크로RNA들이 집중적으로 ‘억제 가능함’을 발견했다. 이는 박지영 생명과학부 교수와 공동연구해 실험으로도 검증됐다.
남덕우 교수는 “이번에 발견한 것은 유방암에서 miR-29가 저하돼 PI3K/Akt 신호전달 경로를 ‘조절하고 있지 않음’”이라며 “miR-29의 발현을 높여주면 해당 경로의 타깃 유전자들과 경로 활성도가 현저히 저하되는 걸 실험적으로 확인했다”고 밝혔다.
또한 미만성 거대 B세포 림프종이라는 질병의 발달을 억제하는 마이크로RNA도 예측해내 이 기법을 다른 여러 질병으로 확장할 수 있음을 보였다.
남 교수는 “BiMIR 데이터베이스를 통해서 누구나 마이크로RNA, 질병 등 세포 조건, 타깃 유전자 등에 대해서 마이크로RNA 조절 네트워크를 검색할 수 있다”며 “현재는 마이크로어레이 데이터 기반으로 만들었는데, RNA 시퀀싱 데이터도 충분해지면 더 다양한 세포 조건에서 더 정확한 네트워크 예측이 가능하다”고 덧붙였다.
이번 연구는 윤소라 생명과학부 박사가 제1저자로 참여했으며, 포스트게놈 다부처유전체사업 및 선도연구센터에서 지원받았다. 연구결과는 영국 옥스퍼드대학 출판사에서 발행하는 저명한 생물학 저널 ‘뉴클레익 에시드 리서치(Nucleic Acids Research, IF: 11.56)’ 3월 1일자 온라인판에 게재됐다.