<편집자주: 과학전문잡지 [과학동아]에서 연재하는 ‘언니오빠 논문연구소’ 코너에 전성원 UNIST 박사과정 연구원의 칼럼이 실렸습니다. 유전체 해독의 길을 제시한 故 프레데릭 생어(Frederick Sanger) 박사의 논문을 통해 유전체 해독 기술을 소개하는 내용입니다.>
유전체는 DNA의 기본단위인 뉴클레오타이드를 이루고 있는 구성 성분인 염기의 조합으로, 모든 생명체가 지니고 있는 ‘생명의 설계도’에 해당합니다. 쉽게 말해 A(아데닌), T(티민), G(구아닌), C(시토신)로 나타낼 수 있는 염기의 서열입니다.
현재 생명과학자들은 인간을 비롯해 다양한 동식물의 유전체 지도를 밝히고 있고, 또 그 안에 유전자가 얼마나 있는지, 각 유전자가 어떤 기능을 하는지 연구하고 있습니다. 그런데 우리가 이렇게 유전체를 해독하게 된 지는 사실 40여 년밖에 되지 않았습니다.
DNA 염기서열을 어떻게 확인할까?
1866년 오스트리아 유전학자인 그레고어 멘델이 완두콩의 형질 유전에 관한 실험으로 ‘멘델의 유전법칙’을 발표하면서 인류는 최초로 유전에 대해 알게 됐습니다. 하지만 그때까지만 해도 어떤 물질이 유전되는 것인지 알지 못했습니다. 이후 1944년 미국 세균학자인 오즈월드 에이버리가 DNA가 유전물질임을 밝혀냈고, 1953년 영국 케임브리지대 제임스 왓슨과 프랜시스 크릭은 DNA가 이중나선 구조라는 사실을 확인했습니다.
당시 왓슨, 크릭과 함께 케임브리지대 분자생물학연구소 소속이었던 프레데릭 생어 박사는 1960년대 말부터 DNA를 해독할 방법을 연구했습니다. 지금처럼 유전체를 빠르고 정확하게 해독할 수 있는 것은 그의 연구 업적에서 시작됐다고 볼 수 있습니다.
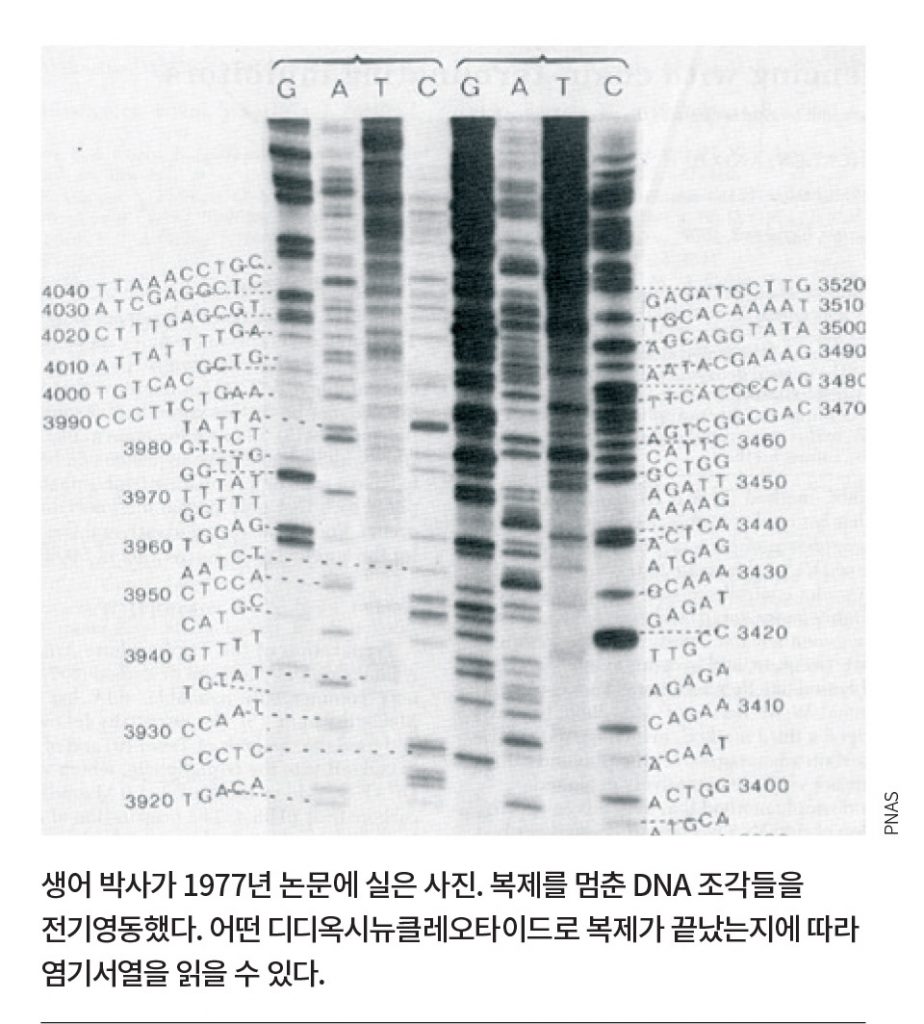 1977년 12월, 생어 박사는 DNA의 염기서열을 해독할 수 있는 핵심 기술에 대한 논문을 국제학술지 ‘미국국립과학원회보(PNAS)’에 발표했습니다. DNA는 DNA 중합효소를 이용해 상보적인 염기끼리 연결시키면서 긴 사슬을 이어나가는 방식으로 복제합니다. 가령 A는 T와 C는 G와 상보적인 염기입니다.
1977년 12월, 생어 박사는 DNA의 염기서열을 해독할 수 있는 핵심 기술에 대한 논문을 국제학술지 ‘미국국립과학원회보(PNAS)’에 발표했습니다. DNA는 DNA 중합효소를 이용해 상보적인 염기끼리 연결시키면서 긴 사슬을 이어나가는 방식으로 복제합니다. 가령 A는 T와 C는 G와 상보적인 염기입니다.
생어 박사는 디디옥시뉴클레오타이드(ddATP, ddGTP, ddCTP, ddTTP)를 이용해 DNA가 복제되는 동안 어느 시점에서 더 이상 DNA 사슬이 연장되지 않도록 중단시켰습니다. 디디옥시뉴클레오타이드는 3’ 말단에 산소가 하나 없어 더 이상 다른 디디옥시뉴클레오타이드가 와서 붙지 못한다는 점을 이용한 것입니다.
생어 박사는 이렇게 복제가 중단된 DNA 조각에서 디디옥시뉴클레오타이드를 종류별로 구분해 젤 전기영동을 진행했습니다. 이렇게 하면 DNA 조각들을 크기별로 줄 세울 수 있습니다. 그리고 맨 마지막에 위치한 디디옥시뉴클레오타이드를 보고 염기의 순서를 읽었습니다.
생어 박사는 이 기술을 이용해 박테리오파지(세균을 감염시키는 바이러스)의 DNA를 정확하게 해독했습니다. 이를 ‘생어 해독법(Sanger Sequencing)’이라고 부릅니다. 생어 해독법으로는 한 번에 염기 300~1000개를 정확하게 읽을 수 있습니다.
내 유전정보 읽는 데 얼마나 걸릴까?
이후 과학자들은 생어 해독법을 이용해 동식물의 유전체를 해독하기 시작했습니다. 그리고 1990년대 들어서는 궁극적으로 인류가 알고 싶어 하는 인간의 유전체를 해독하고자 했습니다. 인간 유전
체 지도를 완성하는 ‘인간 게놈 프로젝트(Human Genome Project)’를 시작한 것입니다.
영국 생어연구소와 미국 연구자들의 주도로 2003년까지 약 13년간 3조 원을 투입한 끝에 인간 유전체 지도의 초안이 탄생했습니다. 인간이 약 2만 개의 유전자를 갖고 있다는 사실도 이때 처음 밝혀졌습니다. 하지만 당시 기술로는 약 30억 개의 염기쌍으로 이뤄진 인간의 유전체를 일일이 분석하기란 불가능했습니다. 가늠할 수 없이 막대한 비용과 많은 시간이 필요했기 때문입니다.
그래서 과학자들은 더욱 싼 가격에, 그리고 훨씬 빠른 속도로 유전체를 분석할 수 있는 기술을 개발하기 시작했습니다. 그리고 1990년 중후반 조지 처치 미국 하버드대 의대 교수를 비롯한 과학자들의 노력으로 ‘차세대 염기서열 분석(NGS·Next Generation Sequencing)’ 기술이 개발됐습니다.
NGS 기술은 DNA를 무작위로 무수히 많은 짧은 조각으로 자른 뒤, 각 조각의 염기서열을 병렬로 동시에 해독합니다. 그래서 짧은 시간에 많은 양의 유전체를 해독할 수 있습니다. 덕분에 2018년 현재 개인의 유전체를 분석하는 데 단 이틀이면 되고, 비용도 약 100만 원 수준으로 저렴해졌습니다.
NGS 기술에도 한계가 있습니다. 기술적으로 DNA 조각 1000개 이상의 염기서열을 한 번에 읽기는 어렵습니다. 최근에는 정보처리 기술을 이용해 유전체 해독 기술을 계속 업그레이드하고 있습니다. 가령 미국의 퍼시픽바이오, 영국 옥스퍼드나노포어테크놀로지가 개발한 새로운 유전체 해독 기술로는 염기 수천 개를 순식간에 읽어냅니다.
유전체 지도는 어디에 활용할까?
유전체 지도를 활용하면 개인에게 딱 맞는 개인 맞춤의학이 가능해집니다. 가장 잘 알려진 사례는 할리우드 스타 안젤리나 졸리입니다. 졸리는 본인의 유전체를 분석한 결과 유방암과 관련된 유전자 (BRCA1)에 변이가 있다는 사실을 확인했고, 2013년 유방암 발병을 예방하기 위해 유방 절제 수술을 받았습니다.
전 세계 생명과학자들은 다양한 종류의 암과 질병의 원인을 밝히고, 개인에게 적절한 치료법을 제시하기 위해 수많은 사람의 유전체 정보를 수집해 분석하고 있습니다. 미국은 2005년부터 ‘암 게놈 아틀라스(The Cancer Genome Atlas)’ 프로젝트를 시작했습니다. 이 프로젝트를 통해 암 환자들로부터 유전체, 전사체(세포 하나에 들어 있는 모든 RNA), 후성유전체(주변 환경 요인 등으로 후천적으로 변한 유전체) 등 다양한 데이터를 확보해 암을 예방하고 치료하는 데 기여하고 있습니다.
국내에서도 2008년 박종화 당시 한국생명공학연구원 국가생물자원정보관리센터장(현 UNIST 게놈산업기술센터장)이 이끄는 연구팀이 최초로 한국인 유전체(SJK)를 해독한 바 있습니다. 세계에서 다섯 번째 개인 유전체 지도였습니다. 중요한 사실은 이 유전체를 당시 아시아인으로는 유일하게 분석된 중국인의 유전체와 비교한 결과 500만 개 정도에서 유전자 차이가 나타났다는 것입니다. 이는 예상보다 훨씬 많은 양이었습니다. 국가별로 유전자 변이가 다른 만큼 결국 개인 유전체를 해독할 필요가 있음을 시사하는 결과였습니다.
필자가 속한 UNIST 게놈산업기술센터는 2014년 한국인의 유전체를 더욱 정밀하고 정확하게 해독해 세계 최초로 한국인 표준유전체지도(KOREF)를 완성했습니다. 2016년 한국인 41명의 유전체 정보를 추가한 유전체 지도(KOREF_C)도 발표했습니다.
우리 연구팀은 조지 처치 교수와 함께 ‘한국인 개인 유전체 프로젝트(KPGP·Korean Personal Genome Project)’를 진행하기도 했습니다. 한국인이 잘 걸리는 질병을 효율적으로 연구하기 위해 한국인 50명의 유전체 데이터를 분석한 결과와 서양인의 유전체 표준(GRCh37)을 비교했습니다. 그 결과는 2018년 4월 국제학술지 ‘사이언티픽 리포트’에 공개됐습니다. (doi:10.1038/s41598-018-23837-x)
현재는 한국인 1만 명 이상의 유전체를 해독하는 ‘울산 만 명 게놈 프로젝트’를 진행하고 있습니다. 이 프로젝트를 통해 한국인의 유전체 정보와 건강 정보, 생활습관 정보를 모아 한국인을 위한 질병예측모델을 개발할 계획입니다. 유전체 기술을 통해 무병장수의 꿈을 이룰 날을 기대해 봅니다.
글: 전성원 jsw0061@gmail.com
필자는 UNIST 게놈산업기술센터 박사과정 연구원이다. 한국인의 게놈 다양성, 게놈과 표현형질의 연관성, 한국인 표준게놈지도에 대한 연구를 하고 있다.













![[배성철 칼럼]‘AI 의사’의 습격인가, 의료 혁명의 서막인가](https://news.unist.ac.kr/kor/wp-content/uploads/2026/02/AI-doc-190x122.jpg)
![[기고] 에너지 안보와 과학의 원칙 위에 서는 지역의 미래](https://news.unist.ac.kr/kor/wp-content/uploads/2026/02/nuclear-190x122.jpg)
![[안현실칼럼]지역 성장의 새 공식을 쓸 기회다](https://news.unist.ac.kr/kor/wp-content/uploads/2026/02/ULSAN-190x122.jpg)
![[목요칼럼]AI는 일자리를 빼앗는가](https://news.unist.ac.kr/kor/wp-content/uploads/2026/02/ai-jobs-190x122.jpg)


